REPORTImageJによるCBB SDS-Page ゲルの検量線の作成
準備
・ タンパク質SDS-Pageの一次元電気泳動したゲルをCBB染色する
・ イメージスキャナ:透過原稿対応スキャナ セイコーエプソン社製 ES-2000を使用
・ スキャナドライバ:EPSON Scan (Version 3.04J)、もしくは「スキャナ駆動ソフトウェアiMeasure Scan」
【スキャン時の留意点】
「自動露出」をかけないようにする。
そのために、「ドライバによる色補正」をOFFにする。もしくは、自動露出をかけずに[RESET]ボタンを押すこと。
解像度:300dpi
イメージタイプ:16bit GrayScale , ドロップアウトカラー Red
・ 電気泳動ゲル解析ソフトウェア:NIH ImageJ (Version 1.41)
下記URLよりダウンロード
・ImageJフリーウェア//imagej.nih.gov/ij/
1.ImageJにてファイルを開く
2.透過率に換算するために最大値を確認する
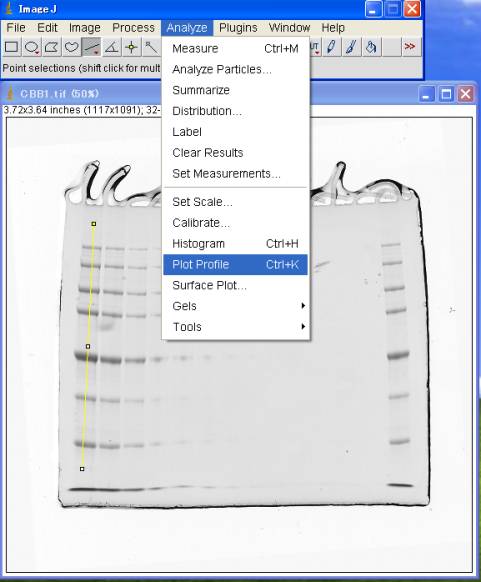
Lineツールにてタンパク質のバンドの部分に線を引く。
Analyze>Plot Profile (Ctrl+K)
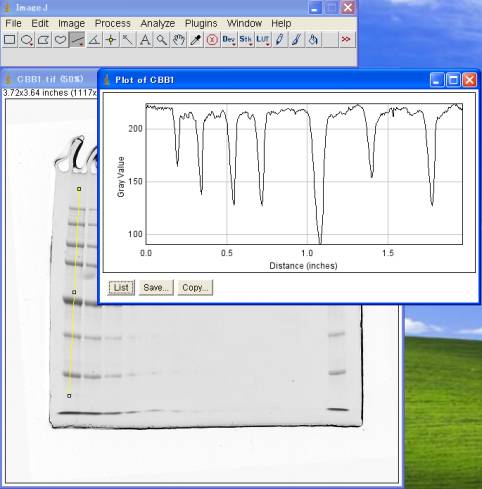
※8bit データの場合 :0~255、16bit データの場合:0~65535
※このファイルは、8bitデータなので、最大値255。
3.イメージタイプを32bitに変更する
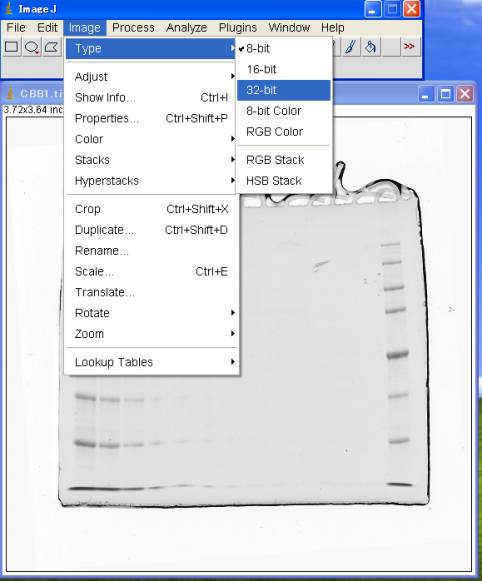
Image>Type>32-bit
※計算誤差を無くすためbit数をあげる。
4.光学濃度に換算する
計算式 【 OD = -1 * (1.8) * ln ( value / 255 ) / ln(10) 】
※EPSON Scanner ドライバのデフォルトガンマは、1.8。
※リニアガンマ(=1.0)でスキャンファイル出力可能であれば1.8は不要。
スキャナ駆動ソフトウェアiMeasure ScanにてDensitometor ONとした場合、スキャナガンマは、1.0となるため計算式の1.8は不要。
詳細は「イメージスキャナ駆動ソフトウェア」を参照。
iMeasure Scan Lite は、vectorからも購入可能となります。
※ImageJの対数計算式は(Logという表記ですが)、Ln(常用対数)です。自然対数にするため、ln(10)で割る。
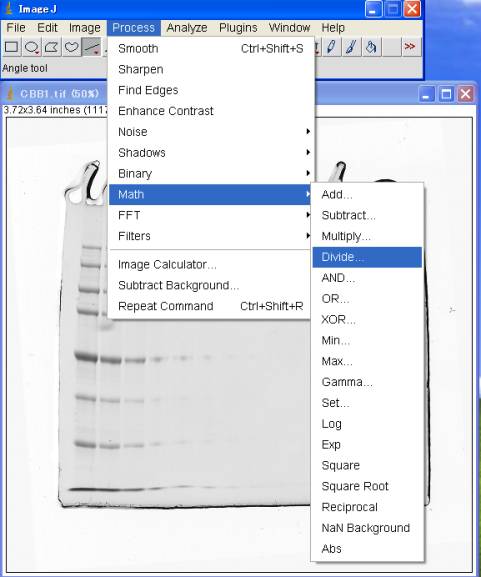
4-1.最大値で割る
Process>Math>Divide 255
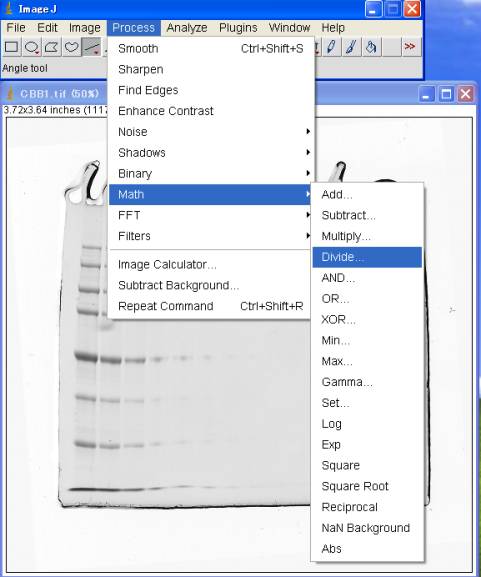
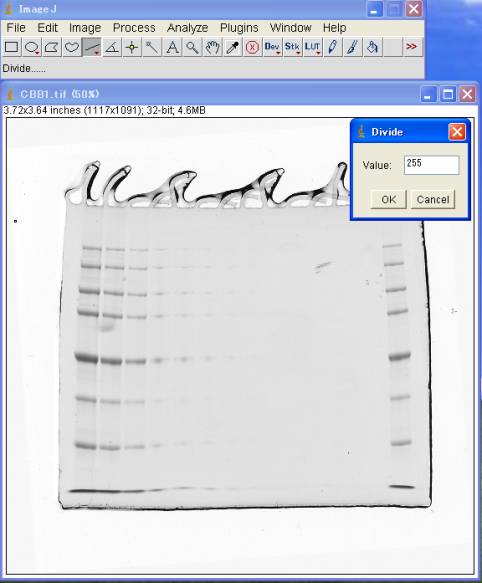
4-2.対数変換する
Process>Math>Log
4-3.Ln対数からLog対数に変換
Ln(10)=2.3025851で割る。
Process>Math>Divide 2.3025851
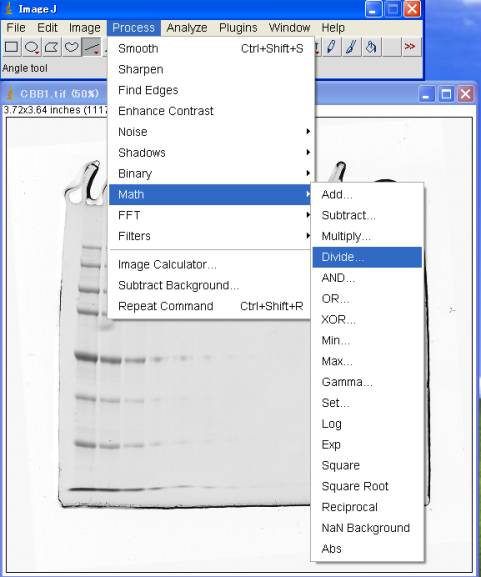
4-4.[1.8]を掛ける
Process>Math>Multiply 1.8
4-5.[-1]を掛ける
Process>Math>Multiply –1
4-6.値を確認する
Lineツールにてタンパク質のバンドの部分に線を引く。
Analyze>Plot Profile (Ctrl+K)
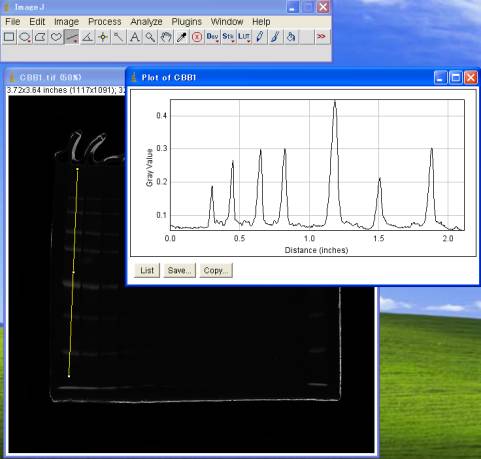
※CBB染色のOD値は、最大でも0.5程度。
5.タンパク質のOD積分値を算出する
画像を見やすくする
Image>Adjust>Brightness/Contrast [Ctrl+Shift+C]
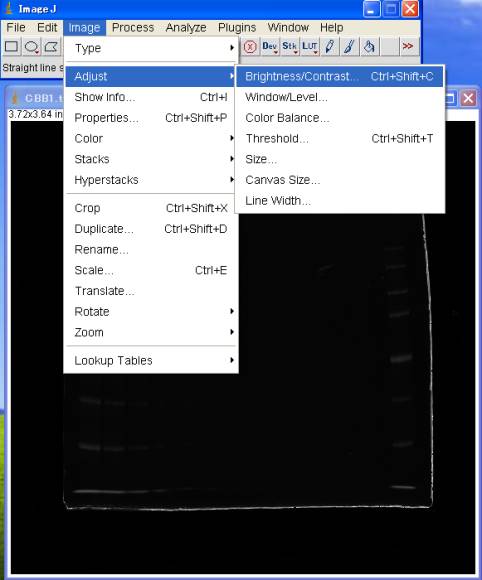
Autoボタンを押す。
B&Cダイアログのヒストグラムを見ながら、Minimum Maximumスライダーを動かして、「ヒストグラムをはさむ」ようにする。
Brightness, Contrastは触らないこと。
※ImageJのBrightness/Contrast による画像操作は、元画像のデータを変更することなく、「見た目」を変えるだけなので、安心して操作する。
※Applyを押してもオリジナル画像のデータは影響を受けない。
5-2.SDS-Page各レーン(バンド)の狙いのタンパクを囲む
今回は、タンパク質分子量 50KDa を計測した。
5-2-1.レーン#1を囲む
Analyze>Gels>Select First Lane (Ctrl+1)
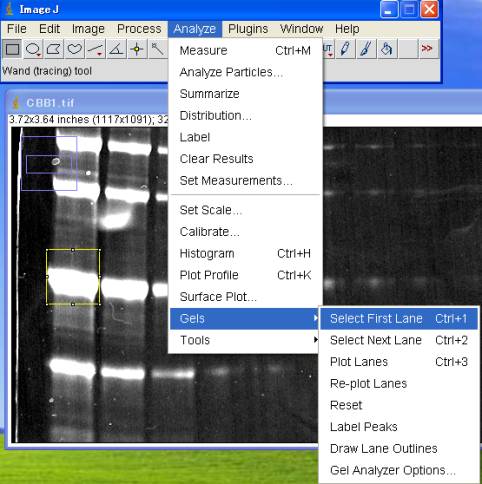
囲った□の中に番号が発生する。
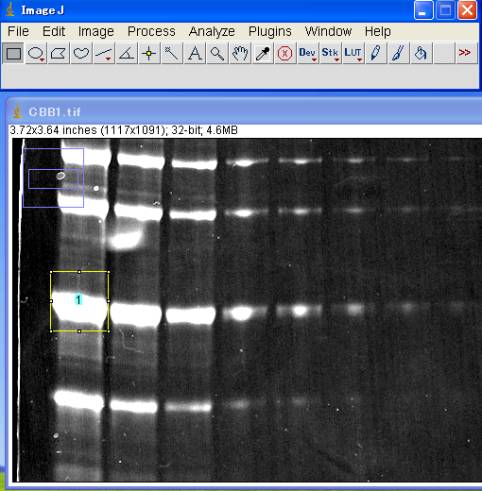
番号の発生した□をブラウスすると、既に複製された□枠が移動する。
5-2-2.レーン#2を囲む
レーン#2の、50kDaのバンドを囲む場所まで移動する。
Analyze>Gels>Select Next Lane (Ctrl+2)
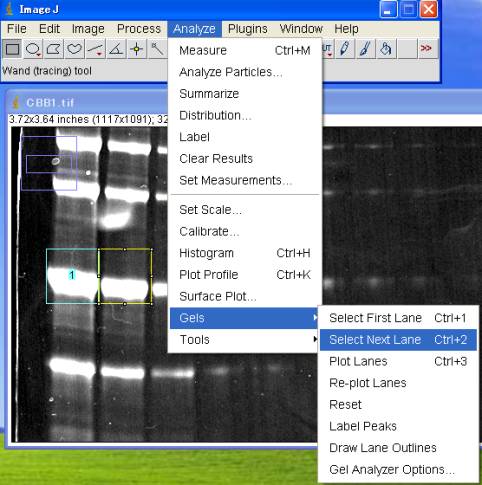
引き続き、レーン#3〜レーン#9まで囲む。
Analyze>Gels>Select Next Lane (Ctrl+2)
5-2-3.ゲル専用の連続[ Plot Profile ]機能を使う
Analyze>Gels>Plot Lanes (Ctrl+3)
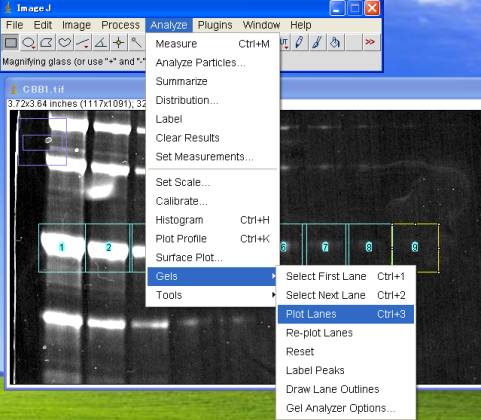
一度に、全レーンの積分濃度のグラフが連続発生する。
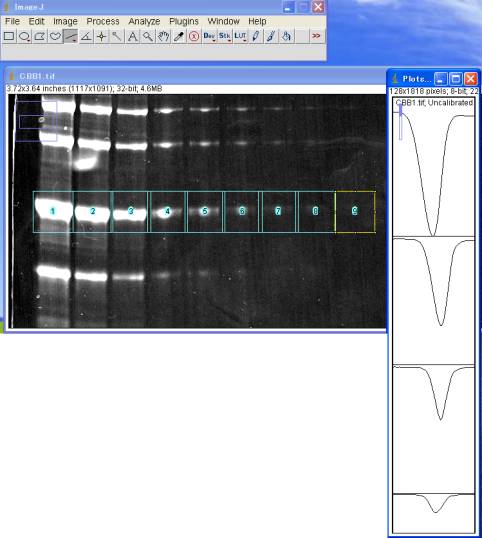
5-3-1.OD積分を算出する
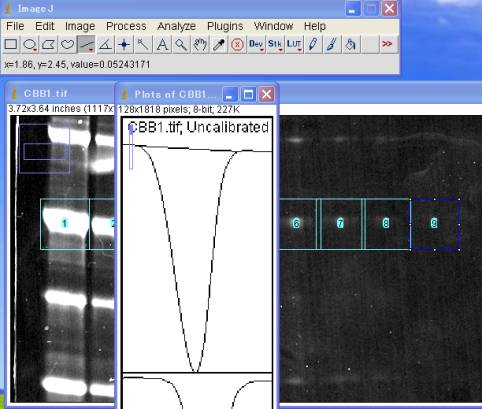
ラインツールを選び、レーン#1のPlot Profile の山のみの積分値を算出するために、ベースラインを引く。
この時、山の内部が、線で囲まれて「空間が閉じた状態」となるように留意する。
失敗したら、キャンセルして線を引き直す。
※ゲル画像上下方向の断面が(Plot Profileにて左右に)表示される。
※ゲル画像左右方向は加算される。
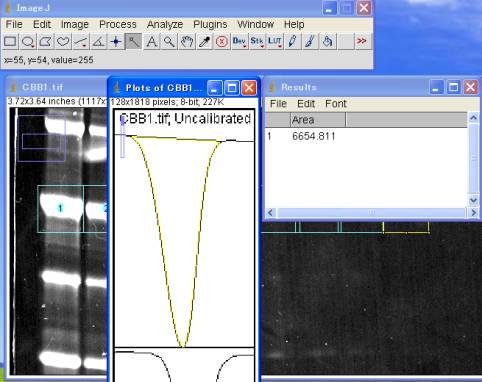
5-3-2.山の面積(OD積分)を算出する
魔法の杖(正式名称は不明?)を選び、山の内部を1回クリックする。
直ちに、Results ダイアログが開き、面積の計算値が表示される。
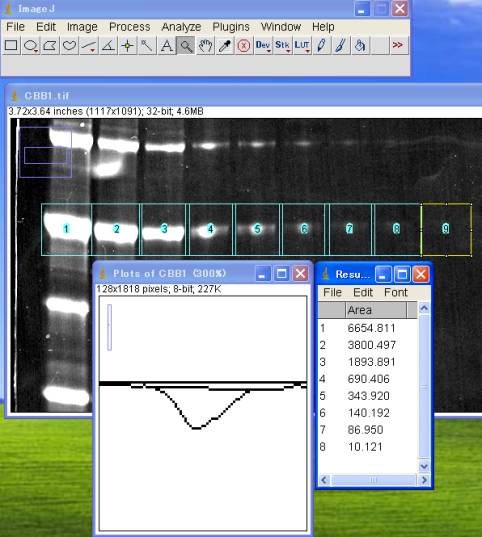
5-3-3.レーン数だけOD積分を算出する
山が次第に小さくなるので、拡大ツールを使って Plots of .. ダイアログを拡大して、山のベースラインを引く。
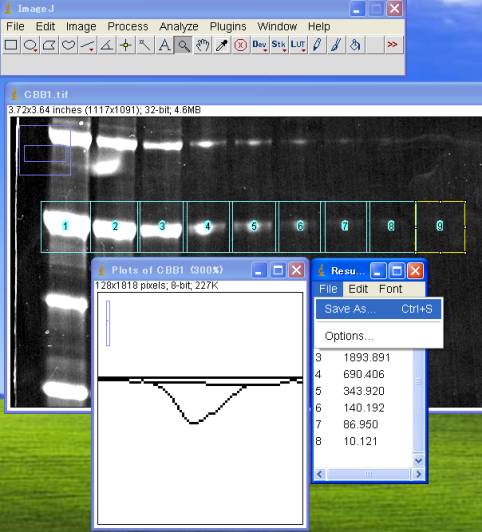
6.ファイル保存する
全てのレーンのOD積分を算出し終えたら、測定値をファイルに保存する。
CSV形式で保存されるので、Excellなどの表計算ソフトで開くことができる。
Result ダイアログ(OD積分値の数値が記載された画面)にて、File>Save As
7.検量線を引く
先に保存したOD積分値ファイルをExcellなどの表計算ソフトで開く。
横軸をゲルの各レーンに投入したタンパク質量 [ng/band]、縦軸に、計測したOD積分値 としてグラフを作成する。
近似式を作成し、相関係数を調べる。
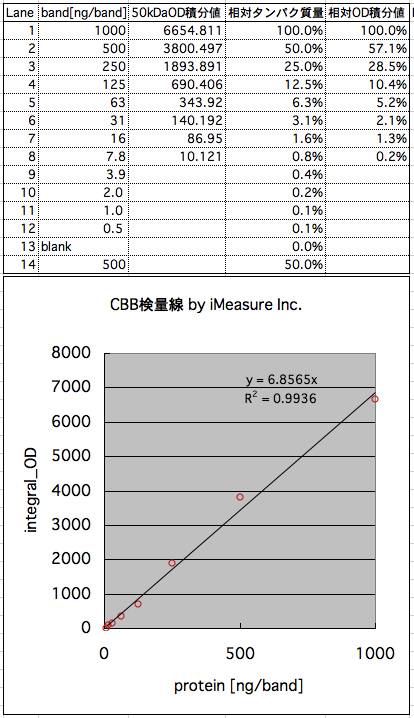
相関係数:0.9936
※相関係数が、1から大きくはずれている場合、次の原因が考えられる。
(1)スキャナの特性が想定外の挙動。
例えば、自動露出が行われていて、勝手にコントラスト強調が行われているなど。
(2)OD値に換算できていない。(Logの計算が抜けている)
OD値ではなく、スキャナの読み値のままの場合、特に高濃度側(グラフ右側)の点が右肩下がりになる。
以上で基本操作は完了です。 お疲れ様でした。
8.検証
対数軸にして表示。
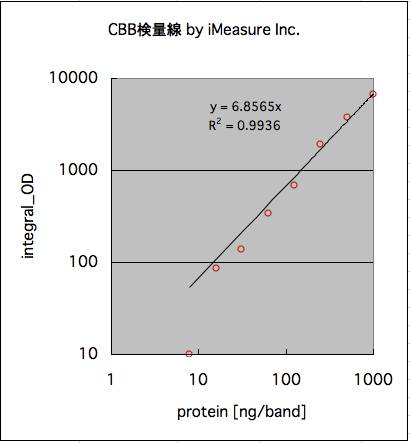
※最も淡いレーン(7.8ng/band)以外は信用できる?
OD積分値は、絶対値ではなく相対値である。
つまり、山の面積を算出する度に異なる値となる。
そこで、タンパク質量の相対比は、繰り返し再現性があるかどうか、チェックした。
実験:レーン#3とレーン#5のみで解析をして、OD積分値の比率を算出。
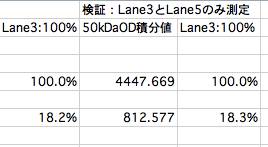
レーン#3の積分値は、先の値(1894)から 4448に変わっているがレーン#3のOD積分値に対するレーン#5のOD積分値は 18.3%であり、先の実験結果 18.2%と精度良く一致する。
 アクセス
アクセス お問合せ
お問合せ